Die passende Antwort auf häufig gestellte Fragen
Finden Sie erklärende Informationen zu unseren Produkten und Dienstleistungen, indem Sie auf die folgenden Links klicken.
Welche Spezies können mit Hilfe der Mikrobiom-Analyse auf dem MiSeq detektiert werden?
Es können alle Bakterien, Archaeen und Pilze identifiziert werden, zu denen eine Sequenz in der NCBI Nukleotid Datenbank publiziert ist. Es gibt momentan zum Beispiel tausende bakterielle Spezies mit Sequenz-Einträgen in der Nukleotid Datenbank. Nah verwandte Arten mit fast identischen DNA-Sequenzen können nicht unterschieden werden. In diesen Fällen wird nur der Genus (oder die Familie) bestimmt.
Welche Einschränkungen gibt es bei der Verwendung von NGS für die Mikrobiom-Analyse?
- Nah verwandte Spezies können aufgrund ihrer fast identischen Sequenz nicht sicher voneinander unterschieden werden.
- Sehr starkt prozessierte Proben oder Proben mit einem sehr geringen pH enthalten zum Teil degradierte DNA. Dies macht eine Speziesbestimmung sehr schwierig und in manchen Fällen unmöglich.
- Je mehr Ausgangsmaterial wir erhalten, desto höher ist die Wahrscheinlichkeit gute Sequenzierergebnisse zu erzielen.
Wie hoch ist der Anteil an falsch positiven oder falsch negativen Ergebnissen?
Bei Proben mit stark degradierter DNA kann es sein, dass keine "Speziesidentifkation" möglich ist. Um falsch posistive Ergebnisse zu vermeiden, wird immer eine Negativkontrolle (Wasser) parallel zu den eigentlichen Proben prozessiert.
Mittels NGS kann jedes DNA Amplikon, das erfolgreich amplifiziert wird auch detektiert werden. Aufgrund der hohen Sensitivität der Methode, müssen Sequenzen, die nur sehr selten in der Probe vorkommen (low coverage) oder Sequenzen mit unerwarteter Sequenzlänge in der Datenanalyse entfernt werden, um falsch positive Ergebnisse, die durch PCR oder Sequenzierfehler entstanden sind, auszuschließen. Spezies, die durch weniger als 0,5% aller Reads abgedeckt werden, werden somit nicht in den PDF Report aufgenommen.
Wie ist die Lieferzeit der Mikrobiom-Analyse?
Die Lieferzeit hängt von der Anzahl der Proben ab. Sie beginnt bei 12 Tagen für bis 192 Proben. Zusätzliche Services, wie die DNA-Extraktion oder die bioinformatische Auswertung verlängern die Lieferzeit. Detaillierte Lieferzeit finden Sie in den Paketbeschreibungen im Bestell-Prozess.
Wie sollte ich die Auswahl an Target-Regionen treffen?
Wählen Sie eines oder mehrere 16S Targets aus um die bakterielle Zusammensetzung in Ihrer Probe zu analysieren, und wählen Sie eines oder beide ITS Targets aus, um die Pilze in Ihrer Probe zu klassifizieren. Für die Analyse von Archaeen wählen Sie bitte das zugehörige 16S Target aus der Liste aus.
Keine der 16S oder ITS Primer Kombination ermöglicht die Amplifikation von allen Spezies. Es ist daher zu empfehlen, mehrere Primer Kombination zu verwenden um die komplette Mikrobiom Population zu analysieren (z.B. Ihrmark et al., 2012; Toju et al., 2012)
Welche Qualitätskontrolle führen Sie bei meinen Proben durch?
Die PCR-Produkt Generierung dient uns als Qualitätskontrolle. Die PCR Bedingungen sind optimiert und der Erfolg der Amplikongenerierung ist von der Menge an bakterieller, archaeller oder Pilz-DNA in der Probe abhängig. Falls kein oder nur ein sehr schwaches Amplikon generiert werden kann, liegt dies in den meisten Fällen an einer zu geringen DNA-Menge. Standardmäßig wiederholen wir die Amplikongenerierung einmalig mit modifizierten Bedingungen in diesen Fällen.
Was passiert wenn Sie kein oder nur ein sehr schwaches Amplikon generieren können?
Wenn Eurofins Genomics bei einigen Ihrer Proben kein Amplikon erzeugen kann, dann werden wir die Bearbeitung dieser Proben einstellen und Ihnen die nicht erbrachten Sequenzierleistungen rückvergüten. Falls ein schwaches Amplikon generiert werden kann, dann wird Eurofins Genomics dieses in den Amplikonpool für die Sequenzierung mit aufnehmen. Erfahrungsgemäß werden für solche Amplikons auch nur wenige Reads generiert. Die Interpretation dieser Reads sollte mit besonderer Vorsicht erfolgen.
Kann ich Ersatzproben schicken?
Es ist nicht möglich Ersatzproben für einen bestehenden Auftrag zu senden. Falls Sie weitere Proben schicken möchten, dann platzieren Sie bitte einen neuen Auftrag. Beide Aufträge werden unabhängig voneinander bearbeitet.
Werden meine Proben mit den Proben anderer Kunden auf einem Lauf sequenziert?
Ja. Ihre Proben werden gemeinsam mit den Proben anderer Kunden auf einem MiSeq Run sequenziert.
Werden Sie meine Proben nach der Prozessierung aufbewahren?
Ihre Proben werden unter geeigneten Bedingungen für 3 Monate nach der Analyse aufbewahrt. Im Anschluss werden die Proben entsorgt, falls keine gesonderten Absprachen vorliegen. Bitte beachten Sie, dass Eurofins Genomics möglicherweise das komplette Probenmaterial für die Analyse verwendet (abhängig davon wieviel Material Sie zu Verfügung stellen können).
Wenn wir das Microbiome Produkt - eigene Targets auswählen, können wir dann Amplikonproben einschicken, die aus mehreren verschiedenen Amplikons, generiert mit verschiedenen Primerpaaren bestehen?
Normalerweise würden Sie pro Probe 1 Amplikon senden, das mit 1 Primerpaar erzeugt wurde. In einigen Fällen können jedoch weniger Reads pro Amplikon erforderlich sein, wie in der Projektspezifikation angegeben. In solchen Fällen können die einzelnen Amplikonproben, die Sie senden, auch aus mehreren Amplikons bestehen, die mit verschiedenen Primerpaaren generiert wurden. In diesem Fall werden wir jedoch keine Primersequenzen clippen und Rohdaten versenden.
Wie soll ich meine Proben verschicken?
Bitte senden Sie aufgereinigte DNA/RNA bzw. Amplikons in 1,5 ml SnapCap Tubes (ohne Parafilm). Und kleben Sie den kostenfreien NGS Barcode quer auf das Tube.
Ab 92 Proben können Sie diese auch gerne in Platten schicken. Bitte verwenden Sie hierfür "Full-Skirted" PCR Platten (zBsp von Eppendorf oder Twin).
Für mehr Informationen und den Probenversand für Extraktionsproben lesen Sie auch unseren Sample Submission Guide.
(Im Bild werden Prepaid Sanger Barcodes verwendet, aber der Ablauf ist für die kostenfreien NGS Barcodes (blau) derselbe)
|
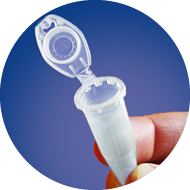
1
Only use 1.5 ml
safe-lock tubes
|

2
Remove barcode
stricker from film
|
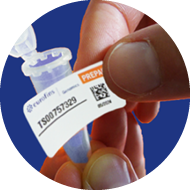
3
Affix barcode sticker
horizontally
|
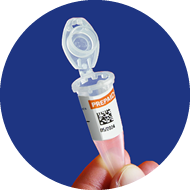
4
QR-code visible at front so
it can be read by scaner
|